6.5 Ejemplo con Datos demográficos¶
Warning
Este cuaderno está realizando con código R
Datos: censo irlandés, area de Dublín, año 2011
Algoritmo SOM en R
Visualización de los resultados
Los códigos R y los datos originales están en el repositorio GIT
Los datos cocinados en: dataIrelandPopulationSOM.csv
Carga y descripción de los datos¶
require(kohonen)
Loading required package: kohonen
data<-read.csv("./data/dataIrelandPopulationSOM.csv")
typeof(data)
head(data,10)
| X | id | avr_age | avr_household_size | avr_education_level | avr_num_cars | avr_health | rented_percent | unemployment_percent | internet_percent | single_percent | married_percent | separated_percent | divorced_percent | widow_percent | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| <int> | <chr> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | <dbl> | |
| 1 | 15687 | 267123023 | 40.02811 | 2.524752 | 3.038462 | 1.0396040 | 4.385542 | 6.930693 | 15.343915 | 71.00000 | 53.41365 | 33.33333 | 4.8192771 | 2.8112450 | 5.6224900 |
| 2 | 13895 | 267016001 | 35.67366 | 3.320611 | 3.597701 | 1.9837398 | 4.509434 | 4.878049 | 12.461059 | 72.95082 | 49.41725 | 44.98834 | 1.3986014 | 0.2331002 | 3.9627040 |
| 3 | 13896 | 267016002 | 35.88235 | 3.324324 | 4.295302 | 1.9054054 | 4.596639 | 1.351351 | 10.404624 | 83.78378 | 47.47899 | 43.69748 | 3.3613445 | 0.0000000 | 5.4621849 |
| 4 | 13729 | 267002034 | 38.51667 | 3.088608 | 3.871795 | 1.7307692 | 4.530172 | 3.896104 | 8.108108 | 78.94737 | 47.08333 | 48.33333 | 0.8333333 | 1.2500000 | 2.5000000 |
| 5 | 13724 | 267002029 | 24.67800 | 3.512000 | 3.933735 | 1.1120000 | 4.510345 | 20.800000 | 21.810700 | 81.30081 | 67.12018 | 26.98413 | 2.2675737 | 1.5873016 | 2.0408163 |
| 6 | 13739 | 267002044 | 25.13953 | 3.136364 | 4.496855 | 1.1090909 | 4.419162 | 29.357798 | 24.154589 | 81.90476 | 64.24419 | 30.23256 | 1.4534884 | 3.4883721 | 0.5813953 |
| 7 | 13725 | 267002030 | 25.05882 | 3.441176 | 4.726872 | 1.5588235 | 4.689888 | 23.529412 | 8.680556 | 94.11765 | 55.55556 | 36.81917 | 3.7037037 | 2.8322440 | 1.0893246 |
| 8 | 13710 | 267002015 | 24.43733 | 2.861538 | 3.973510 | 0.9692308 | 4.580556 | 4.687500 | 19.457014 | 81.25000 | 70.40000 | 21.33333 | 2.9333333 | 3.4666667 | 1.8666667 |
| 9 | 13712 | 267002017 | 23.50530 | 2.958763 | 4.674242 | 1.3917526 | 4.685921 | 20.618557 | 14.044944 | 91.75258 | 62.54417 | 32.86219 | 2.4734982 | 1.7667845 | 0.3533569 |
| 10 | 13713 | 267002018 | 24.67355 | 2.928571 | 4.530769 | 1.4047619 | 4.579832 | 14.814815 | 10.897436 | 82.92683 | 66.52893 | 29.75207 | 1.6528926 | 1.6528926 | 0.4132231 |
names(data)
- 'X'
- 'id'
- 'avr_age'
- 'avr_household_size'
- 'avr_education_level'
- 'avr_num_cars'
- 'avr_health'
- 'rented_percent'
- 'unemployment_percent'
- 'internet_percent'
- 'single_percent'
- 'married_percent'
- 'separated_percent'
- 'divorced_percent'
- 'widow_percent'
dim(data)
- 4806
- 15
Selección de datos y tratamiento¶
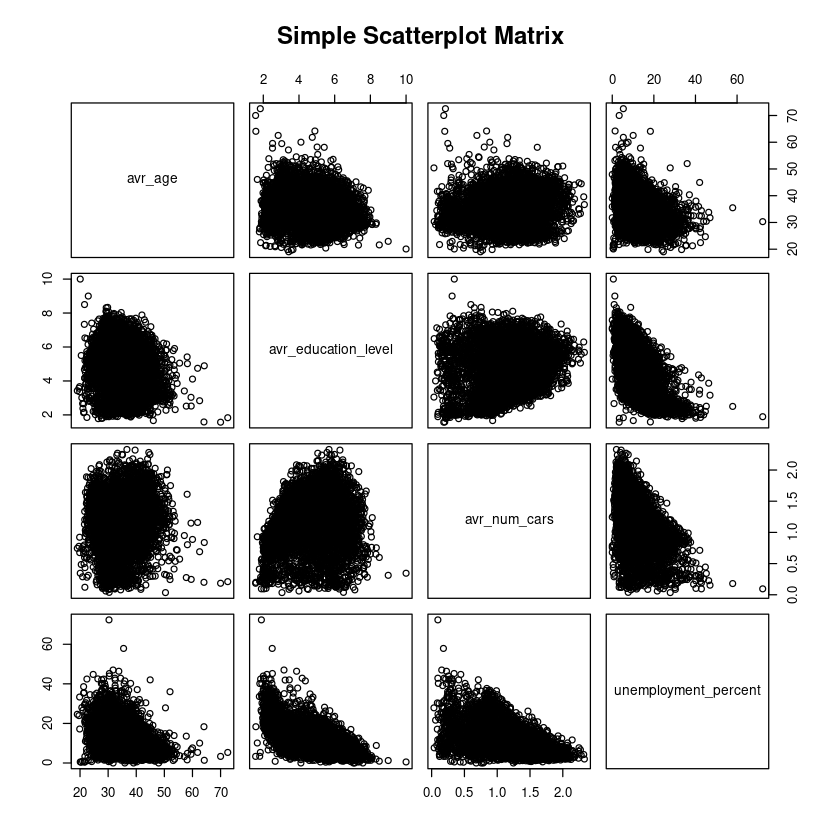
Hacemos una subselección de las variables al conjunto 2,4,5,8.
Centramos y escalamos las variables para que tengan igual importancia durante el proceso SOM
Convertimos los datos a una matriz
c(2,4,5,8)+1
- 3
- 5
- 6
- 9
names(data)[c(2,4,5,8)+1]
- 'avr_age'
- 'avr_education_level'
- 'avr_num_cars'
- 'unemployment_percent'
data_train <- data[,c(2,4,5,8)+1]
head(data_train,10)
| avr_age | avr_education_level | avr_num_cars | unemployment_percent | |
|---|---|---|---|---|
| <dbl> | <dbl> | <dbl> | <dbl> | |
| 1 | 40.02811 | 3.038462 | 1.0396040 | 15.343915 |
| 2 | 35.67366 | 3.597701 | 1.9837398 | 12.461059 |
| 3 | 35.88235 | 4.295302 | 1.9054054 | 10.404624 |
| 4 | 38.51667 | 3.871795 | 1.7307692 | 8.108108 |
| 5 | 24.67800 | 3.933735 | 1.1120000 | 21.810700 |
| 6 | 25.13953 | 4.496855 | 1.1090909 | 24.154589 |
| 7 | 25.05882 | 4.726872 | 1.5588235 | 8.680556 |
| 8 | 24.43733 | 3.973510 | 0.9692308 | 19.457014 |
| 9 | 23.50530 | 4.674242 | 1.3917526 | 14.044944 |
| 10 | 24.67355 | 4.530769 | 1.4047619 | 10.897436 |
data_train_matrix <- as.matrix(scale(data_train))
#''''''
pairs(~avr_age+avr_education_level+avr_num_cars+unemployment_percent,data=data,
main="Simple Scatterplot Matrix")
Definir y entrenar el SOM¶
Creamos el SOM grid con topología hexagonal, y tamaño \(20\times20\).
Entrenamos el SOM usando el grid anterior. Usamos las opciones por defecto del paquete.
som_grid <- somgrid(xdim = 20, ydim=20, topo="hexagonal")
som_model <- som(data_train_matrix,
grid=som_grid,
rlen=500,
alpha=c(0.05,0.01),
keep.data = TRUE )
Visualizar los datos¶
coolBlueHotRed <- function(n, alpha = 1) {
rainbow(n, end=4/6, alpha=alpha)[n:1]
}
pretty_palette <- c("#1f77b4", '#ff7f0e', '#2ca02c', '#d62728', '#9467bd', '#8c564b', '#e377c2')
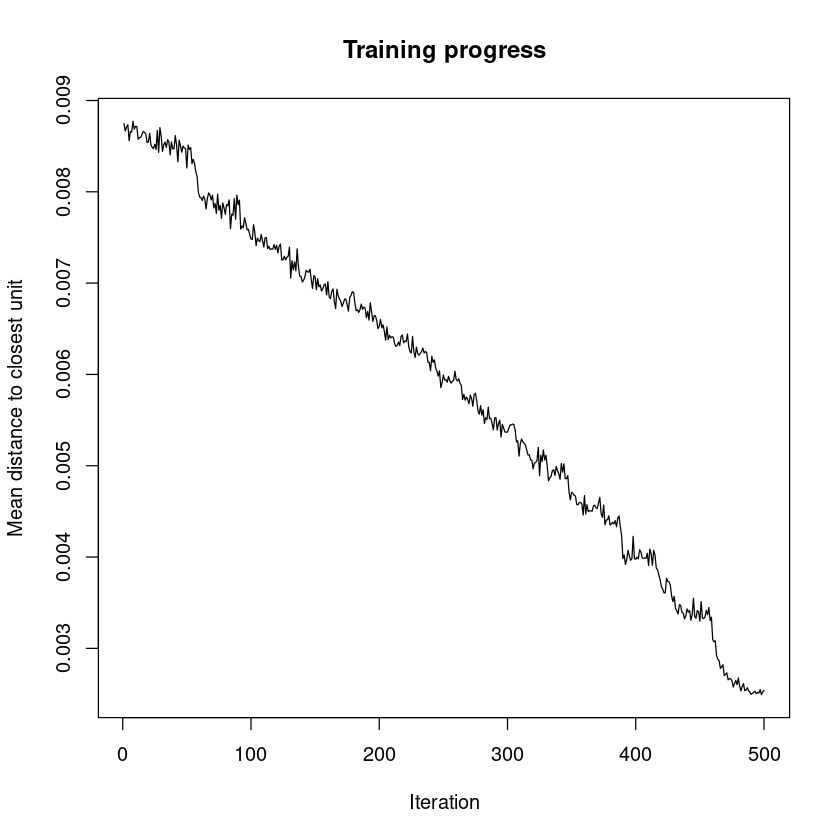
Progreso del entrenamiento¶
A medida que avanza el entrenamiento, la distancia de los pesos de cada nodo a los inputs asociados a dicho nodo se reduce. Idealmente esta distancia debería alcanzar un plateau mínimo.
Si la curva sigue decreciendo quizás son necesarias más iteraciones o cambiar los parámetros de las funciones de aprendizaje y núcleo de vecindad.
plot(som_model, type = "changes")
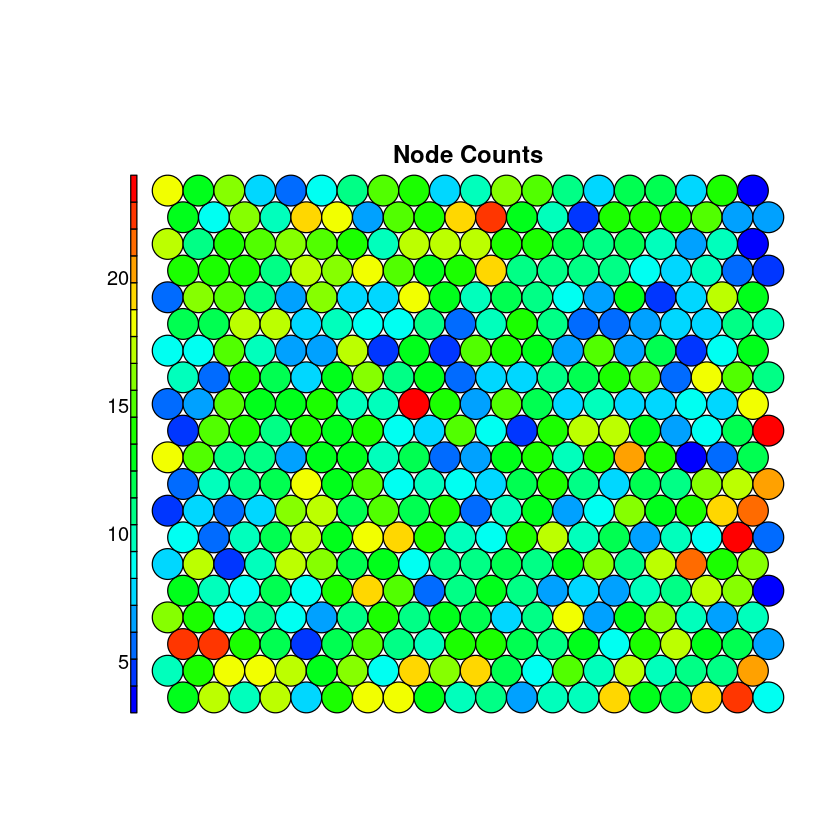
Conteo por nodo¶
Visualizar el número de datos de entrada que están asociados a cada nodo en el mapa.
Esto puede ser usado como una medida de la calidad del mapa:
valores altos en ciertas áreas del mapa sugieren que un mapa más grande puede ser adecuado
muchos nodos vacíos indican que el mapa es demasiado grande
plot(som_model, type = "counts", main="Node Counts", palette.name=coolBlueHotRed)
plot(som_model, type="mapping", main = "Mapeo de puntos", palette.name=coolBlueHotRed)

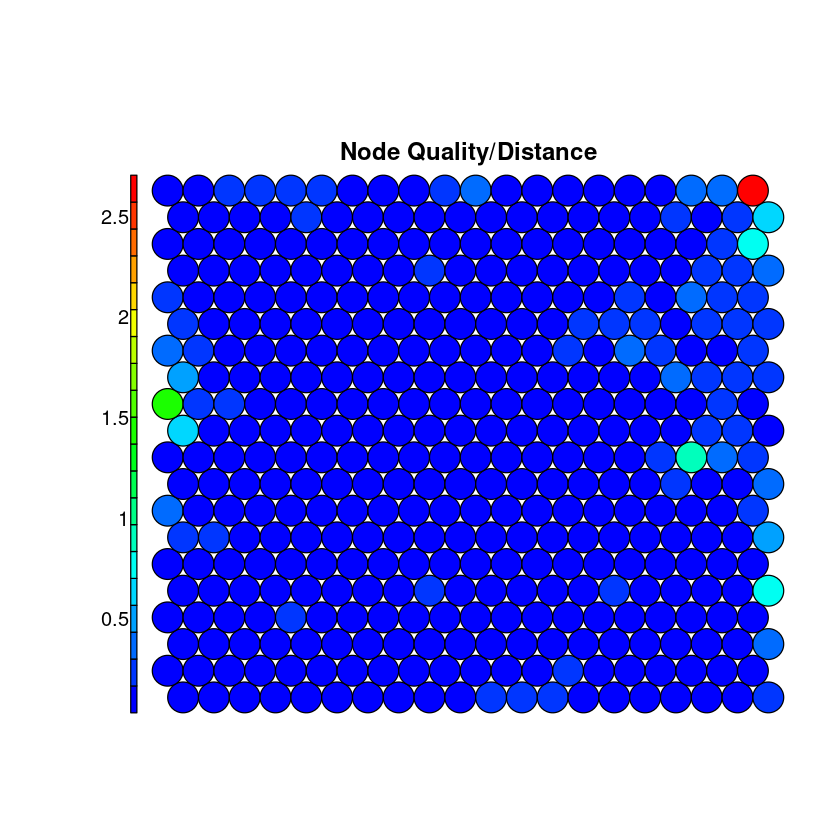
plot(som_model, type = "quality", main="Node Quality/Distance", palette.name=coolBlueHotRed)
U-matrix¶
La \(U\)-matrix es una representación de las distancias entre los nodos.
Áreas donde la distancia entre vecinos sea baja indican grupos de nodos que son similares.
Áreas con grandes distancias entre nodos marcan diferencias e indican fronteras naturales entre los clusters de nodos.
plot(som_model, type="dist.neighbours", main = "SOM neighbour distances", palette.name=grey.colors)

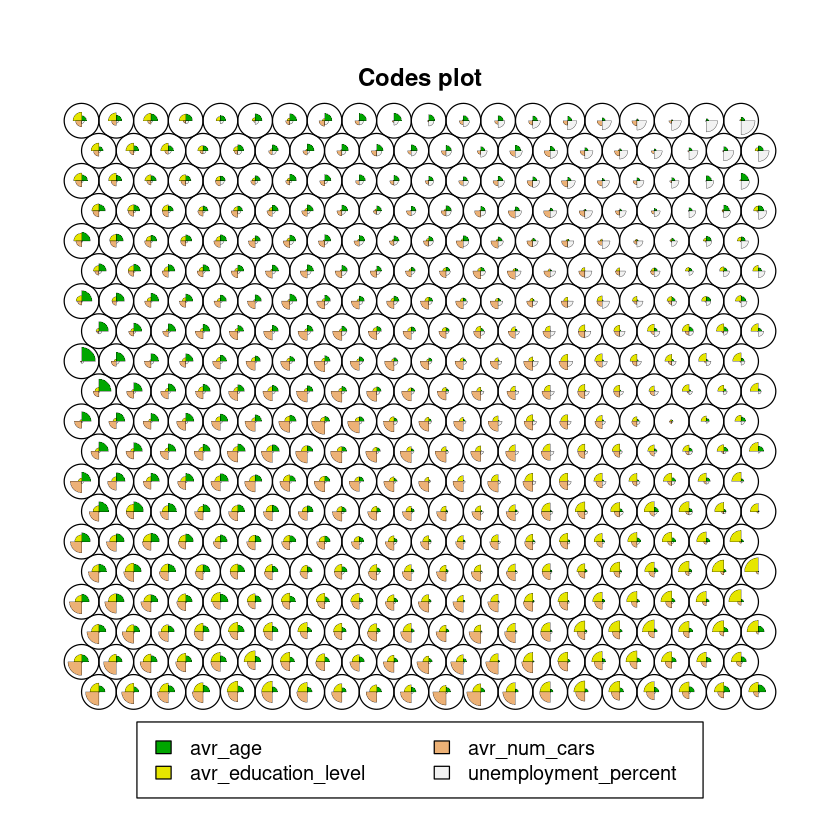
Mápa de códigos¶
En el siguiente mapa, para cada nodo, se representa un diagrama con información de su vector prototipo. En este caso son valors normalizados de los valores originales y, por tanto, se pueden comparar.
Cada vector de pesos de cada nodo es representativo (similar) a los de los datos que están asociados a dicho nodo.
Esta visualización permite enconrtar patrones en la distribución de muestras y variables.
Existe varias opciones para realizar esta presentación.
plot(som_model, type = "codes")
Warning message in par(opar):
“argument 1 does not name a graphical parameter”
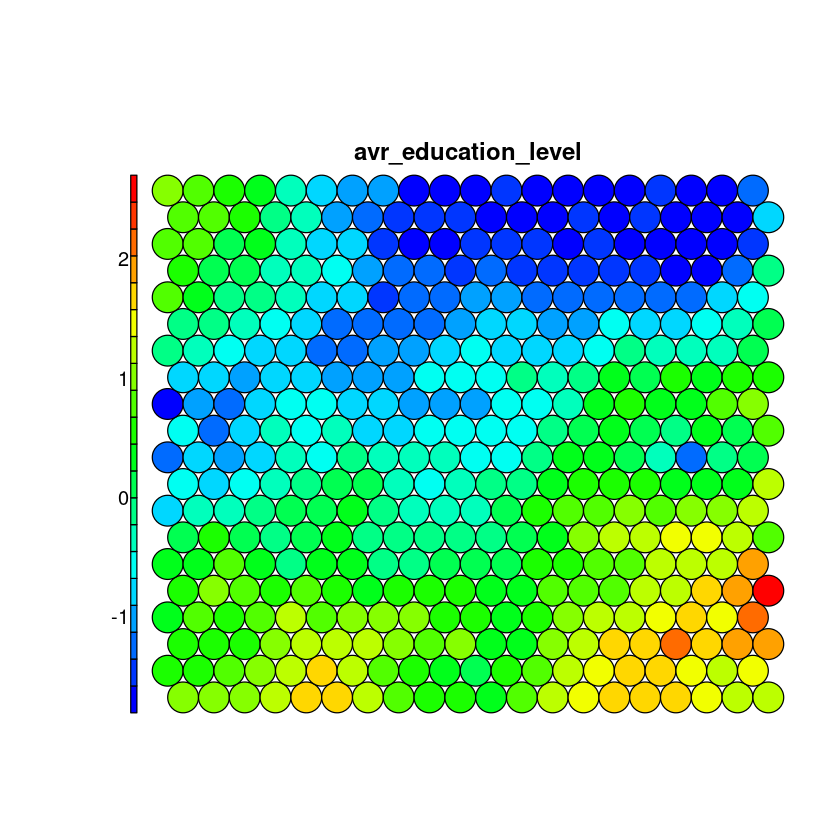
Mapas de calor: heatmaps¶
Los mapas de calor adquieren una especial relevancia en los SOM. El enfoque anterior de presentar los mapas de códigos para cada nodo se vuelve inviable para datos en alta dimensión.
Un mapa de calor permite visualizar la distribución de una variable sobre el mapa.
Recordatorio: los datos de entrada están fijados a su nodo correspondiente, no se mueven. Lo que visualizamos es la distribución de cada uno de sus atributos en el mapa.
var <- 2
plot(som_model,
type = "property",
property = getCodes(som_model)[,var],
main=colnames(getCodes(som_model))[var],
palette.name=coolBlueHotRed
)
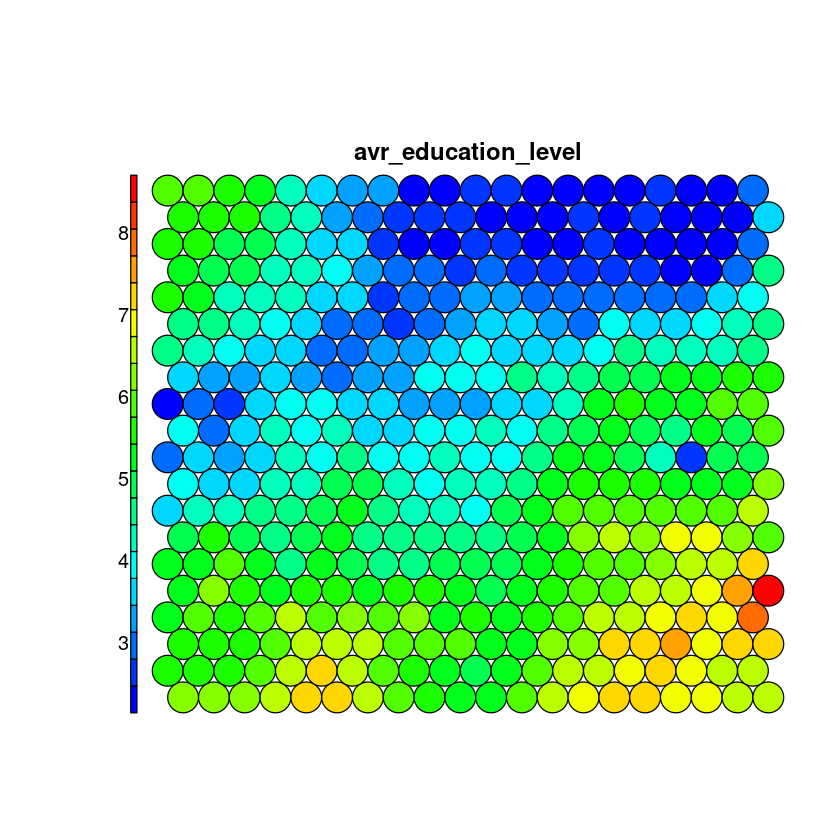
La visualización anterior es de la versión normalizada de la variable de interés. Podemos desnormalizarla.
var <- 2
var_unscaled <- aggregate(as.numeric(data_train[,var]), by=list(som_model$unit.classif), FUN=mean, simplify=TRUE)[,2]
plot(som_model, type = "property", property=var_unscaled, main=names(data_train)[var], palette.name=coolBlueHotRed)
rm(var_unscaled)
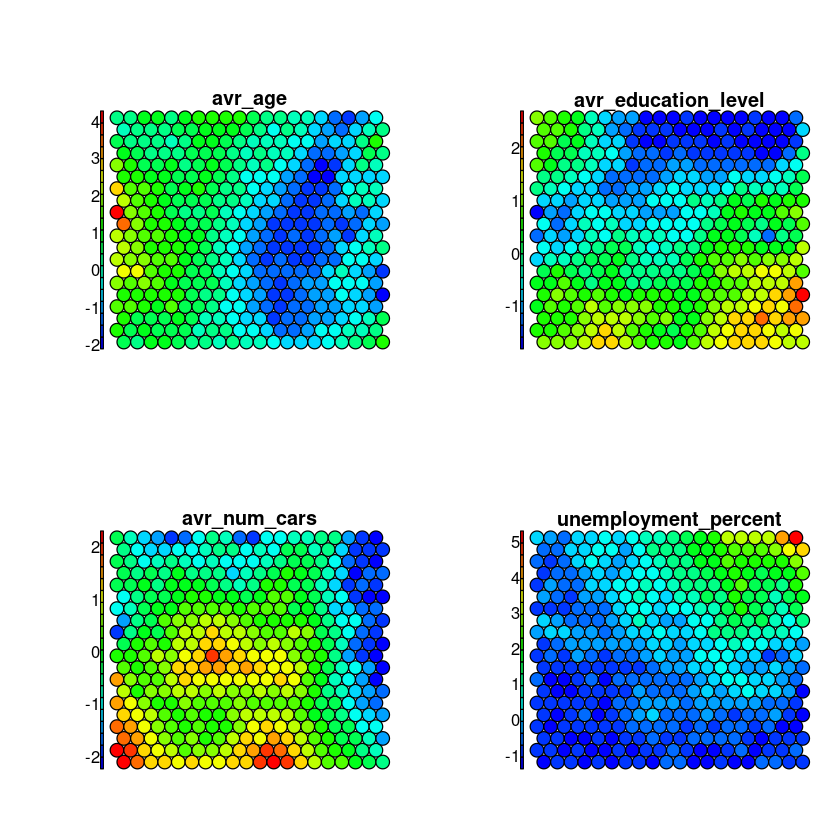
Podemos hacer varios mapas de calor y acumularlos para compararlos visualmente
par(mfrow=c(2,2))
for(var in c(1,2,3,4)){
plot(som_model,
type = "property",
property = getCodes(som_model)[,var],
main=colnames(getCodes(som_model))[var],
palette.name=coolBlueHotRed
)
}
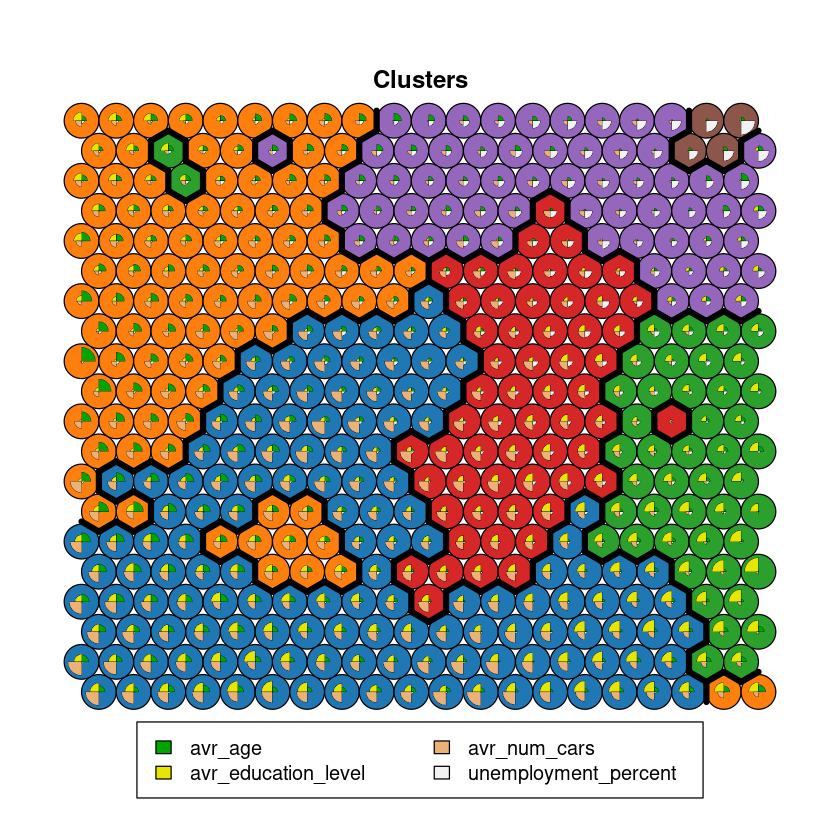
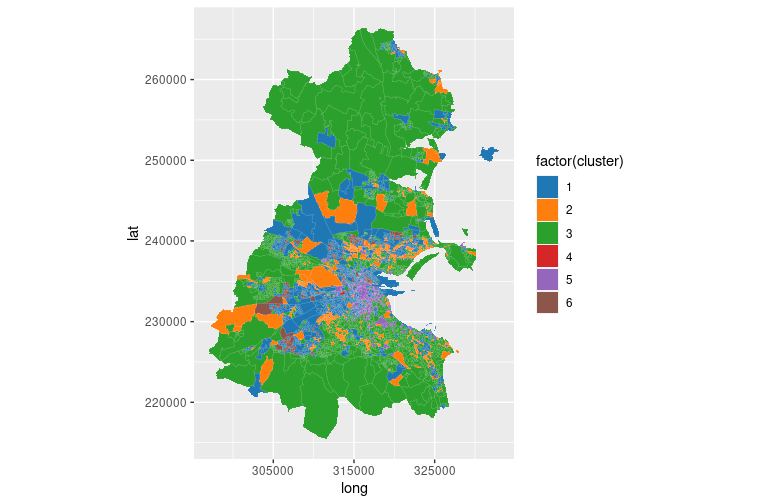
Clustering de los resultados¶
Podemos realizar un algoritmo de clustering sobre el conjunto de los vectores prototipo de los nodos.
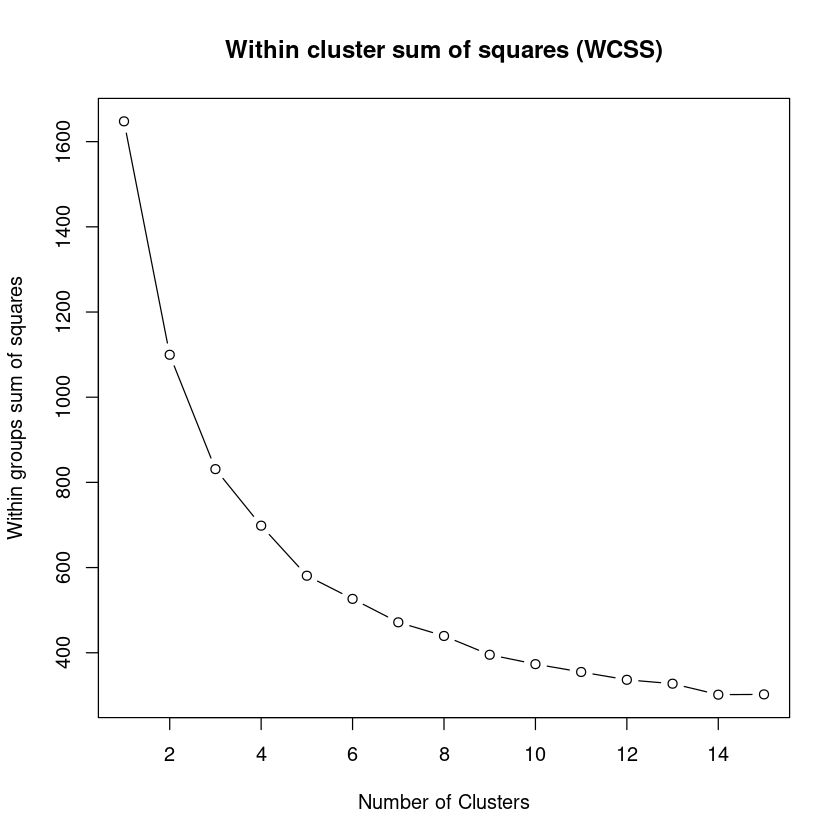
Realizamos una gráfica para averiguar el número de clusters a través de sucesivas aplicaciones de \(k-means\) usando la métrica dada por el WCSS
Después realizamos un hierarchical clustering hclust con el número óptimo que nos indica la gráfica.
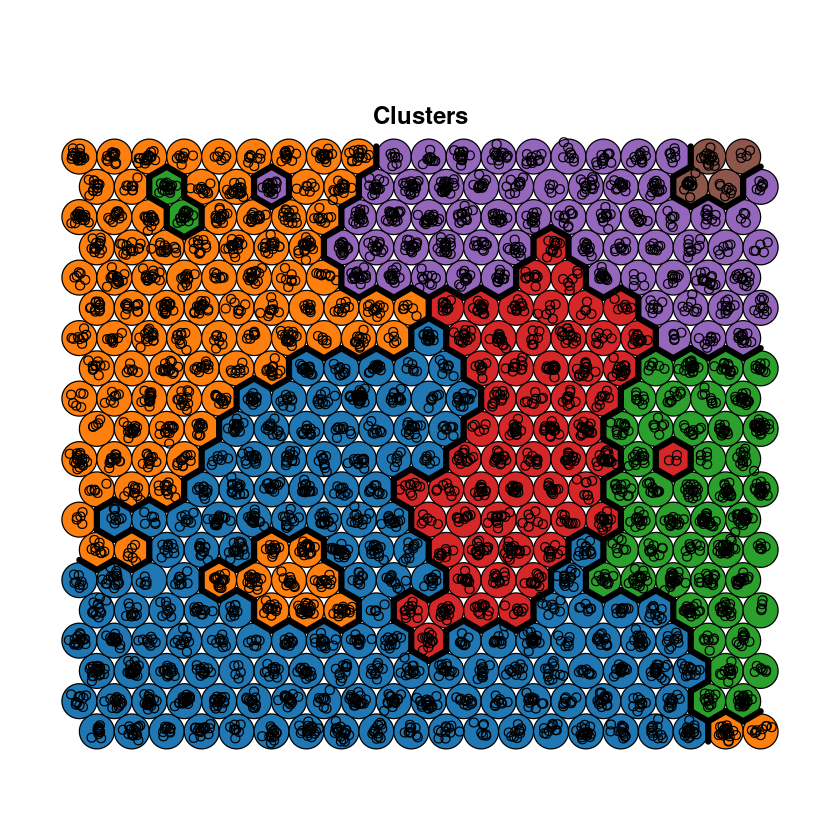
Añadimos finalmente la información de los clusters al mapa SOM.
mydata <- getCodes(som_model)
wss <- (nrow(mydata)-1)*sum(apply(mydata,2,var))
for (i in 2:15) wss[i] <- sum(kmeans(mydata,
centers=i)$withinss)
par(mar=c(5.1,4.1,4.1,2.1))
plot(1:15, wss, type="b", xlab="Number of Clusters",
ylab="Within groups sum of squares", main="Within cluster sum of squares (WCSS)")
som_cluster <- cutree(hclust(dist(getCodes(som_model))), 6)
plot(som_model, type="mapping", bgcol = pretty_palette[som_cluster], main = "Clusters")
add.cluster.boundaries(som_model, som_cluster)
plot(som_model, type="codes", bgcol = pretty_palette[som_cluster], main = "Clusters")
add.cluster.boundaries(som_model, som_cluster)
Warning message in par(opar):
“argument 1 does not name a graphical parameter”