02.2B Clasificación naive-Bayes de fronteras intron-exón o exón-intrón de un conjunto de secuencias de DNA (SOLUCIÓN)¶
“Corta-pega” en RNA:¶
En algunos genes, las secciones del DNA que codifican proteínas (exones) a veces están interrumpidas por regiones no codificantes (intrones).
El proceso de “corta-pega” del RNA quita los intrones del mRNA para generar la secuencia final que codifica la proteina, dejando únicamente los exones en el RNA editado. Los intrones vienen caracterizados por un sitio donor (frontera exón-intrón) y uno aceptor (frontera intrón-exón).
Una animación del proceso se muestra en: https://dnalc.cshl.edu/resources/3d/rna-splicing.html
Problema¶
¿a partir de una lista de secuencias de DNA de las que conocemos “sitios donores”, ¿podemos predecir los sitios donores en nuevas secuencias?
Conjunto de datos de entrenamiento¶
El conjunto de datos esta en el archivo ‘splice.txt’ que puedes descargar de Moodle. Las primeras líneas del archivo son (hemos truncado las ultimas posiciones de las secuencias solo para ajustarlas al texto de este documento):
import numpy as np
import pandas as pd
df = pd.read_csv('data/splice.txt')
df.head()
| Class | Instance | Sequence | |
|---|---|---|---|
| 0 | EI | ATRINS-DONOR-521 | CCAGCTGCATCACAGGAGGCCAGCGAGCAGGTCTGTTCCAAGGGCC... |
| 1 | EI | ATRINS-DONOR-905 | AGACCCGCCGGGAGGCGGAGGACCTGCAGGGTGAGCCCCACCGCCC... |
| 2 | EI | BABAPOE-DONOR-30 | GAGGTGAAGGACGTCCTTCCCCAGGAGCCGGTGAGAAGCGCAGTCG... |
| 3 | EI | BABAPOE-DONOR-867 | GGGCTGCGTTGCTGGTCACATTCCTGGCAGGTATGGGGCGGGGCTT... |
| 4 | EI | BABAPOE-DONOR-2817 | GCTCAGCCCCCAGGTCACCCAGGAACTGACGTGAGTGTCCCCATCC... |
Todas las secuencias tienen 60 pares de bases. La primera columna (Class) indica la clase a la que pertenece la secuencia: EI para las que proporcionan un salto exon-intrón (donores); IE para las secuencias que contienen una frontera intron-exón (aceptores); N para aquellas secuencias que no son ni EI ni IE.
La segunda columna (Instance) es una etiqueta para cada una de las instancias (filas), y la tercera columna da la secuencia correspondiente en terminos del alfabeto A, T, C y G.
Preproceso de las secuencias¶
Algunas de las secuencias tienen ambiguedad en determinadas posiciones, de forma que incluyen las letras D, N, S o R de acuerdo con la siguiente tabla:
Letra |
Significado |
|---|---|
D |
A o G o T |
N |
A o G o T o C |
S |
C o G |
R |
A o G |
Estos caracteres ambiguos aparecen en muy pocas instancias, así que se van a eliminar de los datos para simplificar el analisis. Primero se realiza un proceso de filtrado para saber cuantas secuencias quedan:
df['Sequence'].str.contains("D|N|S|R").value_counts()
False 3175
True 15
Name: Sequence, dtype: int64
df[df['Sequence'].str.contains("D|N|S|R")]
| Class | Instance | Sequence | |
|---|---|---|---|
| 107 | EI | HUMALPI1-DONOR-42 | CACACAGGGCACCCCCTCANNNNNNNNNNNNNNNNNNNNNNNNNNN... |
| 239 | EI | HUMCSPB-DONOR-2010 | AACTTCTCCAACGACATCATGCTACTGCAGGTNAGGCACACTCCTG... |
| 365 | EI | HUMH19-DONOR-2562 | TCTGGGCTCCCAGAACCCACAACATGAAAGGTGAGGGNCTTCCTGC... |
| 366 | EI | HUMH19-DONOR-2766 | ACTACCTGACTCAGGAATCGGCTCTGGAAGGTGAGCACCAGCGCTC... |
| 485 | EI | HUMMHCD8A-DONOR-1087 | TCAGCCACTTCGTGCCGGTCTTCCTGCCAGGTCCGCGCGCCGGGTT... |
| 1247 | IE | HUMMHB27B-ACCEPTOR-1202 | DGACGGGGCTGACCGCGGGGGCGGGTCCAGGGTCTCACACCCTCCA... |
| 1440 | IE | HUMSPRO-ACCEPTOR-2841 | ATACCCCTTTTCACTTTCCCCACCTCTTAGGGTARTCAGTACTGGC... |
| 1441 | IE | HUMSPRO-ACCEPTOR-3195 | CCCTCCTAATGCCCACCATCCCGTCCTCAGGGAAASAGTACTGGGA... |
| 1804 | N | HUMC1A1-NEG-3901 | ATGGCCTACATGGGTGTGGGTGCTGCAATTTCCGCTNCGGCAGACA... |
| 2069 | N | HUMFERG2-NEG-1 | GGATCCCTAGTATAACACATTCAGTGTTCCCCNTTTCAGCCCNTTT... |
| 2135 | N | HUMGCB-NEG-2281 | TGTGAACATGCGCTGTGTGCTGCTTGCTTTGGAAACTNGCCTGGGT... |
| 2578 | N | HUMMHB27B-NEG-1201 | GDGACGGGGCTGACCGCGGGGGCGGGTCCAGGGTCTCACACCCTCC... |
| 2636 | N | HUMMHDRR2-NEG-121 | TAAATCCTCTCGCTGCGTGGTGAGAAAACTGATGCCTNGAGTCTGT... |
| 2637 | N | HUMMHDRS1-NEG-241 | AAAATACAAAAATTAGCCNNNGGGAGGCCGAGATTACAATGAGCTG... |
| 2910 | N | HUMRSCNTT-NEG-121 | TGAGGTTAAAGGTNGAAAAGGAAATATCTTCGTATAAAAACTAGAC... |
Finalmente se borran los registros de secuencia ambigua
Nota: Hay que recordar que en una lista de datos con con true se copian y ~(Alt-126 o insertar símbolo) niega.
df = df[~df['Sequence'].str.contains("D|N|S|R")]
df
| Class | Instance | Sequence | |
|---|---|---|---|
| 0 | EI | ATRINS-DONOR-521 | CCAGCTGCATCACAGGAGGCCAGCGAGCAGGTCTGTTCCAAGGGCC... |
| 1 | EI | ATRINS-DONOR-905 | AGACCCGCCGGGAGGCGGAGGACCTGCAGGGTGAGCCCCACCGCCC... |
| 2 | EI | BABAPOE-DONOR-30 | GAGGTGAAGGACGTCCTTCCCCAGGAGCCGGTGAGAAGCGCAGTCG... |
| 3 | EI | BABAPOE-DONOR-867 | GGGCTGCGTTGCTGGTCACATTCCTGGCAGGTATGGGGCGGGGCTT... |
| 4 | EI | BABAPOE-DONOR-2817 | GCTCAGCCCCCAGGTCACCCAGGAACTGACGTGAGTGTCCCCATCC... |
| ... | ... | ... | ... |
| 3185 | N | ORAHBPSBD-NEG-2881 | TCTCTTCCCTTCCCCTCTCTCTTTCTTTCTTTTCTCTCCTCTTCTC... |
| 3186 | N | ORAINVOL-NEG-2161 | GAGCTCCCAGAGCAGCAAGAGGGCCAGCTGAAGCACCTGGAGAAGC... |
| 3187 | N | ORARGIT-NEG-241 | TCTCGGGGGCGGCCGGCGCGGCGGGGAGCGGTCCCCGGCCGCGGCC... |
| 3188 | N | TARHBB-NEG-541 | ATTCTACTTAGTAAACATAATTTCTTGTGCTAGATAACCAAATTAA... |
| 3189 | N | TARHBD-NEG-1981 | AGGCTGCCTATCAGAAGGTGGTGGCTGGTGTGGCTGCTGCTCTGGC... |
3175 rows × 3 columns
Obtener dos subconjuntos, uno de entrenamiento y otro de validación¶
Divide el conjunto de datos en una parte para entrenamiento y otra parte para validacion, empleando muestreo aleatorio sin repetición. Reserva un 70 % de las instancias para entrenamiento y un 30 % para el conjunto de validacion
from sklearn.model_selection import train_test_split
X, y = df.values[:,2], df.values[:,0]
X_train, X_test, y_train, y_test =train_test_split(X, y, train_size=0.7, random_state=0, stratify=y, shuffle=True)
Implementación con la librería sk-learn¶
Primero se prepara la matriz X de los conjuntos de entranamiento y pruebas¶
Para hacer el entrenamiento hay que convertir la cadena de 60 caracteres en 60 caracteristicas de 1 caracter:
X_train60 = [[letra for letra in fila] for fila in X_train]
X_test60 = [[letra for letra in fila] for fila in X_test]
print(len(X_train60), len(X_train60[0]), len(y_train))
print(len(X_test60), len(X_test60[0]), len(y_test))
2222 60 2222
953 60 953
Además se convierten los caracteres alfabéticos en números¶
Hay varias posibilidades para hacer ésto, damos una:
df = pd.DataFrame(X_train60)
bases=['A','T','C','G']
base_mapping = {'A': 0, 'T': 1, 'C': 2, 'G': 3}
for iCol in range(60):
df[iCol] = df[iCol].map(base_mapping)
X_train60 = df.values
df = pd.DataFrame(X_test60)
bases=['A','T','C','G']
base_mapping = {'A': 0, 'T': 1, 'C': 2, 'G': 3}
for iCol in range(60):
df[iCol] = df[iCol].map(base_mapping)
X_test60 = df.values
X_train60.shape, X_test60.shape
((2222, 60), (953, 60))
Se Resuelve utilizando el clasificador Naive Bayes de sklearn¶
from sklearn.naive_bayes import GaussianNB
clf_NB = GaussianNB()
clf_NB.fit(X_train60, y_train)
tot_Test = np.size(y_test)
tot_aciertos = 0
y_pred = []
for x, y in zip(X_test60, y_test):
y_est = clf_NB.predict([x])
y_pred.append(y_est)
if (y==y_est): tot_aciertos +=1
#print('Instancia %s Clase real %s - Clase estimada %s \n' %(x, y, y_est))
print('Exactitud del clasificador (porcentaje de aciertos) = %s \n' %(tot_aciertos*100/tot_Test))
Exactitud del clasificador (porcentaje de aciertos) = 90.97586568730325
Y se presenta la correspondiente matriz de confusión¶
from sklearn.metrics import confusion_matrix
import matplotlib.pyplot as plt
cm=confusion_matrix(y_test, y_pred)
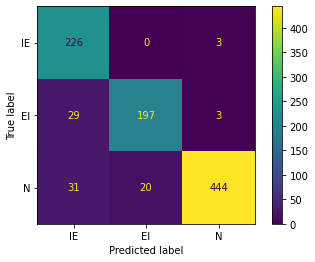
print(cm)
[[226 0 3]
[ 29 197 3]
[ 31 20 444]]
from sklearn.metrics import plot_confusion_matrix
clases=['IE','EI','N']
plot_confusion_matrix(clf_NB, X_test60, y_test, display_labels=clases)
/usr/local/lib/python3.8/dist-packages/sklearn/utils/deprecation.py:87: FutureWarning: Function plot_confusion_matrix is deprecated; Function `plot_confusion_matrix` is deprecated in 1.0 and will be removed in 1.2. Use one of the class methods: ConfusionMatrixDisplay.from_predictions or ConfusionMatrixDisplay.from_estimator.
warnings.warn(msg, category=FutureWarning)
<sklearn.metrics._plot.confusion_matrix.ConfusionMatrixDisplay at 0x7fe4e5f5a7f0>